R言語のrpartを使って決定木を作ってみる
R言語を使って、決定木を作ってみました。 きちんとRを使ったことないのですが、お手軽に出来て出力も綺麗で、結構良い感じです。
データセットにはR言語に標準で付属しているirisというやつを使います。 アヤメの種類と、その計測のデータらしいです。 アヤメの知識は全く無いけれど、アヤメをどうやって分類したら良いか知りたい。というときに、決定木というやつが役に立つようです(?)
R言語ではrpartというライブラリが良い感じにやってくれます。以下のような感じ。
> library(rpart) > rpart(Species ~ ., data=iris) n= 150 node), split, n, loss, yval, (yprob) * denotes terminal node 1) root 150 100 setosa (0.33333333 0.33333333 0.33333333) 2) Petal.Length< 2.45 50 0 setosa (1.00000000 0.00000000 0.00000000) * 3) Petal.Length>=2.45 100 50 versicolor (0.00000000 0.50000000 0.50000000) 6) Petal.Width< 1.75 54 5 versicolor (0.00000000 0.90740741 0.09259259) * 7) Petal.Width>=1.75 46 1 virginica (0.00000000 0.02173913 0.97826087) *
計算させただけですが、この時点でもなんとなく結果が読めてうれしいですね。
でもやっぱり見やすくはないので、きちんとグラフにします。可視化にはpartykitというライブラリが使えるようです。
> library(rpart) > library(partykit) > iris.part <- as.party(rpart(Species ~ ., data=iris)) > iris.part Model formula: Species ~ Sepal.Length + Sepal.Width + Petal.Length + Petal.Width Fitted party: [1] root | [2] Petal.Length < 2.45: setosa (n = 50, err = 0.0%) | [3] Petal.Length >= 2.45 | | [4] Petal.Width < 1.75: versicolor (n = 54, err = 9.3%) | | [5] Petal.Width >= 1.75: virginica (n = 46, err = 2.2%) Number of inner nodes: 2 Number of terminal nodes: 3 > plot(iris.part)
コンソールの時点でかなり見やすくなってうれしい。
プロットしてあげると、以下のようなグラフが表示されます。
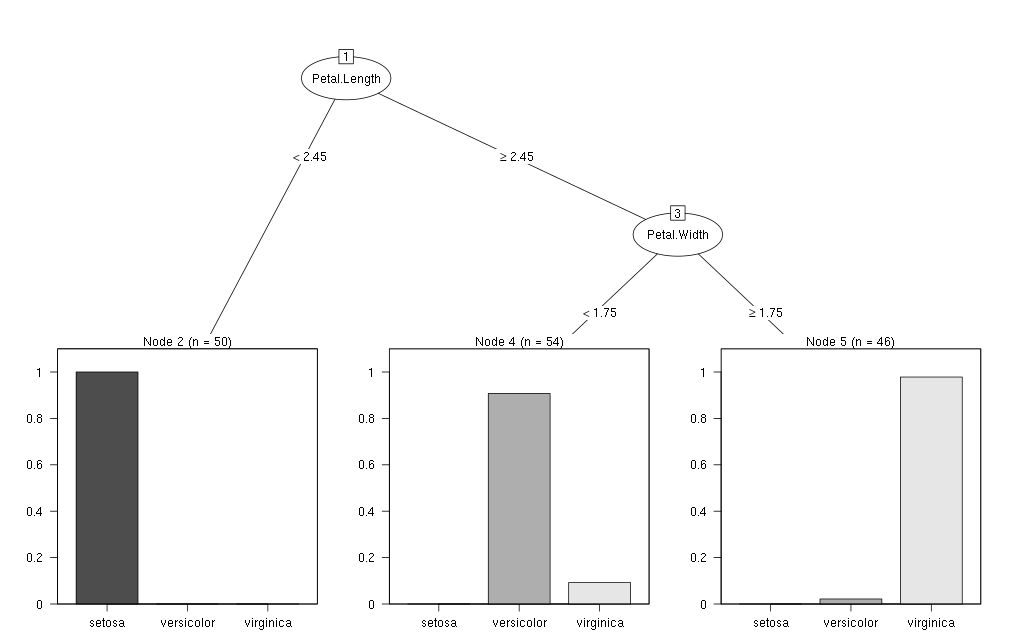
この決定木を見れば、Petal(花弁)の長さが短かいものはsetosaという品種に。長いものについては、花弁の幅を見ればversicolorかvirginicaかを区別出来るのだと分かります。
irisをSVMで分類するようなサンプルをよく見ますが、個人的には決定木で分類した方が特性が分かって良いような気がします。 勿論知りたい情報によって使い分ける必要があると思うので、色々出来るようにならないといけないのでしょうけれど。
